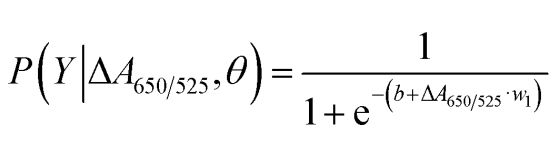Wczesne wykrywanie nowotworów na podstawie biopsji płynnej to nowy kierunek w wykrywaniu i diagnostyce nowotworów, zaproponowany w ostatnich latach przez Narodowy Instytut Raka w USA, mający na celu wykrywanie wczesnych stadiów raka, a nawet zmian przedrakowych. Jest on szeroko stosowany jako nowy biomarker do wczesnej diagnostyki różnych nowotworów złośliwych, w tym raka płuc, nowotworów przewodu pokarmowego, glejaków i nowotworów ginekologicznych.
Powstanie platform służących do identyfikacji biomarkerów metylacji (Methylscape) ma potencjał, aby znacząco ulepszyć istniejące wczesne badania przesiewowe w kierunku raka, umożliwiając pacjentom rozpoczęcie leczenia na najwcześniejszym możliwym etapie.
Niedawno naukowcy opracowali prostą i bezpośrednią platformę pomiarową do wykrywania metylacji opartą na nanocząsteczkach złota zdobionych cysteaminą (Cyst/AuNP) w połączeniu z biosensorem na smartfonie, która umożliwia szybkie i wczesne badania przesiewowe szerokiego spektrum nowotworów. Wczesne badania przesiewowe w kierunku białaczki można przeprowadzić w ciągu 15 minut od ekstrakcji DNA z próbki krwi, z dokładnością 90,0%. Tytuł artykułu: Szybkie wykrywanie DNA nowotworów we krwi ludzkiej z wykorzystaniem nanocząsteczek AuNP pokrytych cysteaminą i smartfona z funkcją uczenia maszynowego.
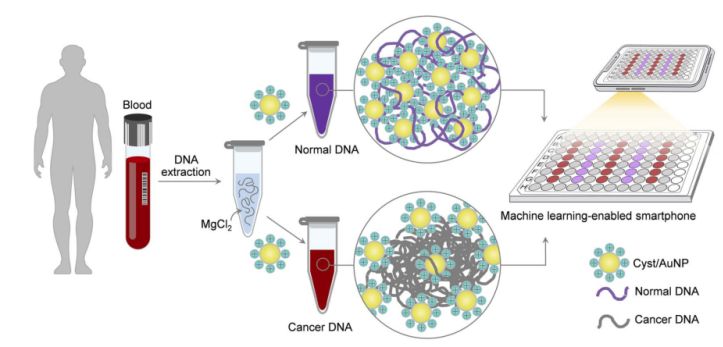
Rysunek 1. Prostą i szybką platformę czujnikową do badań przesiewowych w kierunku raka za pomocą komponentów Cyst/AuNP można zbudować w dwóch prostych krokach.
Przedstawiono to na rysunku 1. Najpierw użyto roztworu wodnego do rozpuszczenia fragmentów DNA. Następnie do mieszaniny dodano nanocząstki cyst/AuNP. Prawidłowe i złośliwe DNA charakteryzują się różnymi właściwościami metylacji, co skutkuje powstaniem fragmentów DNA o różnych wzorcach samoorganizacji. Prawidłowe DNA agreguje luźno, a ostatecznie agreguje nanocząstki cyst/AuNP, co powoduje przesunięcie ku czerwieni nanocząstek cyst/AuNP, dzięki czemu zmianę koloru z czerwonego na fioletowy można zaobserwować gołym okiem. Natomiast unikalny profil metylacji DNA nowotworowego prowadzi do powstawania większych skupisk fragmentów DNA.
Zdjęcia płytek 96-dołkowych wykonano aparatem w smartfonie. Pomiar DNA nowotworowego wykonano za pomocą smartfona wyposażonego w technologię uczenia maszynowego, w porównaniu z metodami opartymi na spektroskopii.
Badania przesiewowe w kierunku raka w rzeczywistych próbkach krwi
Aby rozszerzyć użyteczność platformy pomiarowej, badacze zastosowali czujnik, który skutecznie odróżniał DNA prawidłowe od nowotworowego w rzeczywistych próbkach krwi. Wzorce metylacji w miejscach CpG epigenetycznie regulują ekspresję genów. W niemal wszystkich typach nowotworów zaobserwowano naprzemienne zmiany w metylacji DNA, a tym samym w ekspresji genów promujących nowotworzenie.
Jako model dla innych nowotworów związanych z metylacją DNA, naukowcy wykorzystali próbki krwi pobrane od pacjentów z białaczką i zdrowych osób z grupy kontrolnej, aby zbadać skuteczność analizy metylacji w różnicowaniu nowotworów białaczkowych. Ten biomarker metylacji nie tylko przewyższa istniejące szybkie metody przesiewowe w kierunku białaczki, ale także dowodzi możliwości rozszerzenia go na wczesne wykrywanie szerokiego spektrum nowotworów za pomocą tego prostego i łatwego w użyciu testu.
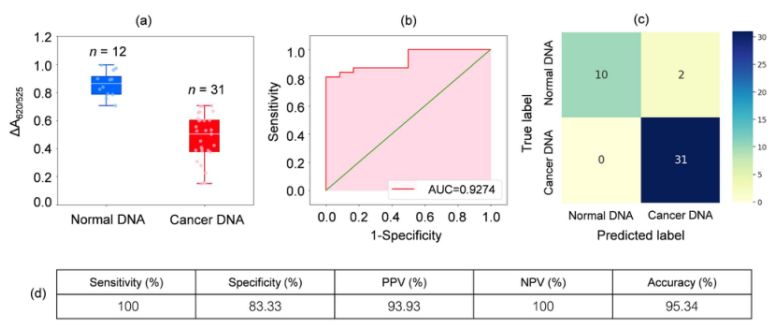
Przeanalizowano DNA z próbek krwi pobranych od 31 pacjentów z białaczką i 12 osób zdrowych. Jak pokazano na wykresie pudełkowym na rysunku 2a, względna absorbancja próbek nowotworowych (ΔA650/525) była niższa niż w przypadku DNA z próbek zdrowych. Wynikało to głównie ze zwiększonej hydrofobowości, prowadzącej do gęstej agregacji DNA nowotworowego, co zapobiegało agregacji Cyst/AuNP. W rezultacie nanocząstki te uległy całkowitemu rozproszeniu w zewnętrznych warstwach agregatów nowotworowych, co skutkowało różną dyspersją Cyst/AuNP zaadsorbowanych na agregatach DNA zdrowych i nowotworowych. Następnie wygenerowano krzywe ROC, zmieniając próg od wartości minimalnej ΔA650/525 do wartości maksymalnej.
Rysunek 2.(a) Wartości względnej absorbancji roztworów cyst/AuNPs pokazujące obecność prawidłowego (niebieskiego) i nowotworowego (czerwonego) DNA w zoptymalizowanych warunkach
(DA650/525) wykresów pudełkowych; (b) Analiza ROC i ocena testów diagnostycznych. (c) Macierz błędów w diagnostyce pacjentów zdrowych i chorych na raka. (d) Czułość, swoistość, dodatnia wartość predykcyjna (PPV), ujemna wartość predykcyjna (NPV) i dokładność opracowanej metody.
Jak pokazano na rysunku 2b, pole pod krzywą ROC (AUC = 0,9274) uzyskane dla opracowanego czujnika wykazało wysoką czułość i swoistość. Jak widać na wykresie pudełkowym, najniższy punkt reprezentujący grupę DNA prawidłowego nie jest wyraźnie oddzielony od najwyższego punktu reprezentującego grupę DNA nowotworowego; dlatego do rozróżnienia grup zdrowych i nowotworowych zastosowano regresję logistyczną. Biorąc pod uwagę zbiór zmiennych niezależnych, regresja ta szacuje prawdopodobieństwo wystąpienia zdarzenia, takiego jak grupa nowotworowa lub zdrowa. Zmienna zależna przyjmuje wartości od 0 do 1. Wynik jest zatem prawdopodobieństwem. Prawdopodobieństwo identyfikacji nowotworu (P) określono na podstawie ΔA650/525 w następujący sposób.
gdzie b = 5,3533, w1 = -6,965. W przypadku klasyfikacji próbki prawdopodobieństwo mniejsze niż 0,5 oznacza próbkę prawidłową, a prawdopodobieństwo 0,5 lub wyższe oznacza próbkę nowotworową. Rysunek 2c przedstawia macierz pomyłek wygenerowaną w wyniku walidacji krzyżowej typu „zostaw to w spokoju”, która została wykorzystana do walidacji stabilności metody klasyfikacji. Rysunek 2d podsumowuje ocenę diagnostyczną metody, w tym czułość, swoistość, dodatnią wartość predykcyjną (PPV) i ujemną wartość predykcyjną (NPV).
Biosensory oparte na smartfonach
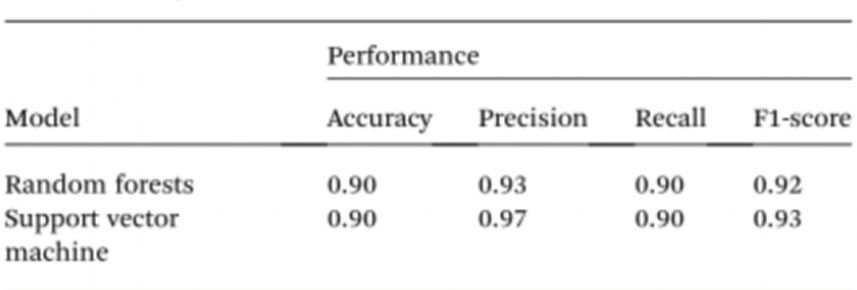
Aby jeszcze bardziej uprościć badanie próbek bez użycia spektrofotometrów, naukowcy wykorzystali sztuczną inteligencję (AI) do interpretacji koloru roztworu i rozróżnienia osób zdrowych od chorych na raka. Biorąc to pod uwagę, zastosowano wizję komputerową do przetłumaczenia koloru roztworu Cyst/AuNPs na normalne DNA (fioletowe) lub nowotworowe DNA (czerwone) za pomocą obrazów płytek 96-dołkowych wykonanych aparatem telefonu komórkowego. Sztuczna inteligencja może obniżyć koszty i poprawić dostępność w interpretacji koloru roztworów nanocząstek, bez użycia jakichkolwiek optycznych akcesoriów do smartfonów. Na koniec wytrenowano dwa modele uczenia maszynowego, w tym model lasu losowego (RF) i model maszyny wektorów nośnych (SVM), aby zbudować modele. Oba modele RF i SVM poprawnie klasyfikowały próbki jako pozytywne i negatywne z dokładnością 90,0%. Sugeruje to, że wykorzystanie sztucznej inteligencji w bioczujnikach opartych na telefonach komórkowych jest całkiem możliwe.
Rysunek 3.(a) Klasa docelowa roztworu zarejestrowana podczas przygotowywania próbki do etapu akwizycji obrazu. (b) Przykładowy obraz wykonany podczas etapu akwizycji obrazu. (c) Intensywność koloru roztworu cyst/AuNP w każdym dołku płytki 96-dołkowej wyodrębnionej z obrazu (b).
Wykorzystując Cyst/AuNP, naukowcy z powodzeniem opracowali prostą platformę pomiarową do wykrywania metylacji oraz czujnik zdolny do odróżniania prawidłowego DNA od DNA nowotworowego podczas badań przesiewowych w kierunku białaczki z wykorzystaniem rzeczywistych próbek krwi. Opracowany czujnik wykazał, że DNA wyekstrahowane z rzeczywistych próbek krwi pozwala na szybkie i ekonomiczne wykrywanie niewielkich ilości DNA nowotworowego (3 nM) u pacjentów z białaczką w ciągu 15 minut, osiągając dokładność 95,3%. Aby dodatkowo uprościć badanie próbek poprzez wyeliminowanie potrzeby stosowania spektrofotometru, wykorzystano uczenie maszynowe do interpretacji koloru roztworu i odróżnienia osób zdrowych od nowotworowych na podstawie zdjęcia wykonanego telefonem komórkowym. Dokładność pomiaru również osiągnęła 90,0%.
Odniesienie: DOI: 10.1039/d2ra05725e
Czas publikacji: 18 lutego 2023 r.
 中文网站
中文网站