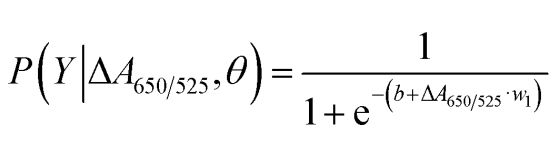Wczesne wykrywanie raka na podstawie płynnej biopsji to nowy kierunek wykrywania i diagnostyki nowotworów zaproponowany w ostatnich latach przez amerykański National Cancer Institute, mający na celu wykrywanie wczesnych zmian nowotworowych, a nawet zmian przedrakowych.Jest szeroko stosowany jako nowy biomarker we wczesnej diagnostyce różnych nowotworów złośliwych, w tym raka płuc, nowotworów przewodu pokarmowego, glejaków i guzów ginekologicznych.
Pojawienie się platform do identyfikacji biomarkerów krajobrazu metylacji (Methylscape) może znacząco poprawić istniejące wczesne badania przesiewowe w kierunku raka, umieszczając pacjentów na najwcześniejszym możliwym do wyleczenia etapie.
Niedawno naukowcy opracowali prostą i bezpośrednią platformę wykrywania do wykrywania krajobrazu metylacji w oparciu o nanocząsteczki złota ozdobione cysteaminą (Cyst/AuNP) w połączeniu z biosensorem opartym na smartfonie, który umożliwia szybkie wczesne badanie przesiewowe szerokiej gamy nowotworów.Wczesne badanie przesiewowe w kierunku białaczki można przeprowadzić w ciągu 15 minut po ekstrakcji DNA z próbki krwi, z dokładnością do 90,0%.Tytuł artykułu to Szybkie wykrywanie nowotworowego DNA w ludzkiej krwi przy użyciu AuNP zakończonych cysteaminą i smartfona z funkcją uczenia maszynowego.
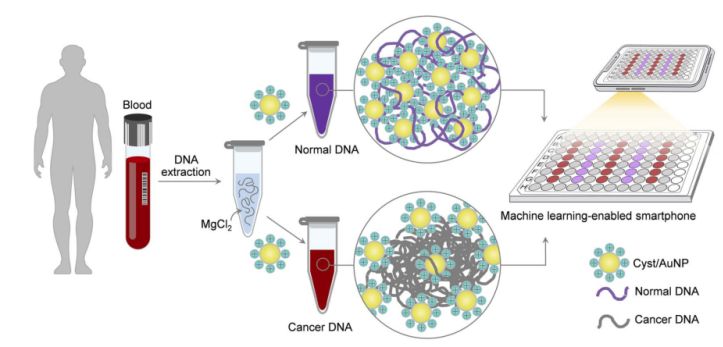
Rycina 1. Prostą i szybką platformę wykrywania raka za pomocą komponentów Cyst/AuNPs można wykonać w dwóch prostych krokach.
Pokazano to na rycinie 1. Najpierw do rozpuszczenia fragmentów DNA użyto roztworu wodnego.Następnie do zmieszanego roztworu dodano Cyst/AuNP.Normalne i złośliwe DNA mają różne właściwości metylacji, w wyniku czego fragmenty DNA mają różne wzorce samoorganizacji.Normalne DNA agreguje luźno i ostatecznie agreguje Cyst/AuNP, co skutkuje przesunięciem ku czerwieni natury Cyst/AuNP, tak że zmianę koloru z czerwonego na fioletowy można zaobserwować gołym okiem.W przeciwieństwie do tego, unikalny profil metylacji nowotworowego DNA prowadzi do wytwarzania większych skupisk fragmentów DNA.
Obrazy 96-dołkowych płytek wykonano za pomocą aparatu w smartfonie.DNA raka zostało zmierzone za pomocą smartfona wyposażonego w uczenie maszynowe w porównaniu z metodami opartymi na spektroskopii.
Badania przesiewowe w kierunku raka w prawdziwych próbkach krwi
Aby rozszerzyć użyteczność platformy czujnikowej, badacze zastosowali czujnik, który z powodzeniem rozróżnia normalne i nowotworowe DNA w prawdziwych próbkach krwi.wzorce metylacji w miejscach CpG epigenetycznie regulują ekspresję genów.W prawie wszystkich typach nowotworów obserwowano naprzemienne zmiany w metylacji DNA, a tym samym w ekspresji genów promujących nowotworzenie.
Jako model dla innych nowotworów związanych z metylacją DNA, naukowcy wykorzystali próbki krwi od pacjentów z białaczką i zdrowych osób z grupy kontrolnej, aby zbadać skuteczność krajobrazu metylacji w różnicowaniu nowotworów białaczkowych.Ten biomarker krajobrazu metylacji nie tylko przewyższa istniejące szybkie metody przesiewowe białaczki, ale także demonstruje wykonalność rozszerzenia na wczesne wykrywanie szerokiego zakresu nowotworów za pomocą tego prostego i bezpośredniego testu.
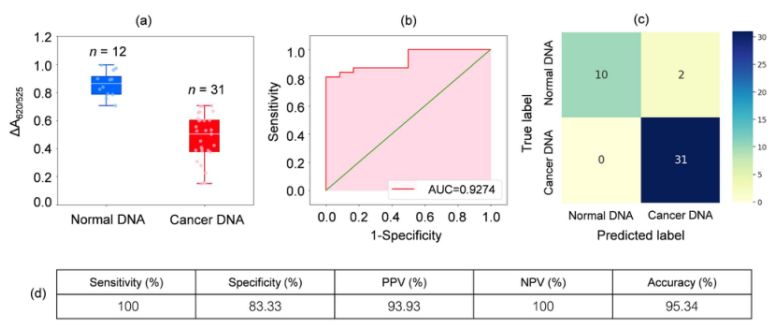
Przeanalizowano DNA z próbek krwi pobranych od 31 pacjentów z białaczką i 12 zdrowych osób.jak pokazano na wykresie pudełkowym na rycinie 2a, względna absorbancja próbek raka (ΔA650/525) była niższa niż absorbancja DNA z normalnych próbek.wynikało to głównie ze zwiększonej hydrofobowości prowadzącej do gęstej agregacji nowotworowego DNA, co zapobiegało agregacji Cyst/AuNP.W rezultacie te nanocząsteczki zostały całkowicie rozproszone w zewnętrznych warstwach agregatów nowotworowych, co skutkowało inną dyspersją Cyst/AuNP zaadsorbowanych na agregatach normalnego i nowotworowego DNA.Następnie wygenerowano krzywe ROC zmieniając próg od wartości minimalnej ΔA650/525 do wartości maksymalnej.
Rycina 2. (a) Względne wartości absorbancji roztworów cyst/AuNPs pokazujące obecność normalnego (niebieskiego) i rakowego (czerwonego) DNA w zoptymalizowanych warunkach
(DA650/525) wykresów pudełkowych;(b) Analiza ROC i ocena testów diagnostycznych.(c) Matryca zamieszania do diagnozy pacjentów zdrowych i chorych na raka.(d) Czułość, specyficzność, dodatnia wartość predykcyjna (PPV), ujemna wartość predykcyjna (NPV) oraz dokładność opracowanej metody.
Jak pokazano na rysunku 2b, pole pod krzywą ROC (AUC = 0,9274) uzyskane dla opracowanego czujnika wykazało wysoką czułość i specyficzność.Jak widać z wykresu pudełkowego, najniższy punkt reprezentujący normalną grupę DNA nie jest dobrze oddzielony od najwyższego punktu reprezentującego grupę nowotworowego DNA;dlatego zastosowano regresję logistyczną do rozróżnienia między grupami normalnymi i nowotworowymi.Biorąc pod uwagę zestaw zmiennych niezależnych, szacuje prawdopodobieństwo wystąpienia zdarzenia, takiego jak rak lub normalna grupa.Zmienna zależna zawiera się w przedziale od 0 do 1. Wynik jest zatem prawdopodobieństwem.Określiliśmy prawdopodobieństwo identyfikacji raka (P) na podstawie ΔA650/525 w następujący sposób.
gdzie b=5,3533, w1=-6,965.W przypadku klasyfikacji próbek prawdopodobieństwo mniejsze niż 0,5 wskazuje na normalną próbkę, podczas gdy prawdopodobieństwo 0,5 lub wyższe wskazuje na próbkę nowotworową.Rycina 2c przedstawia macierz zamieszania wygenerowaną z walidacji krzyżowej typu „zostaw to w spokoju”, która została wykorzystana do sprawdzenia stabilności metody klasyfikacji.Rycina 2d podsumowuje ocenę testu diagnostycznego metody, w tym czułość, swoistość, dodatnią wartość predykcyjną (PPV) i ujemną wartość predykcyjną (NPV).
Bioczujniki oparte na smartfonach
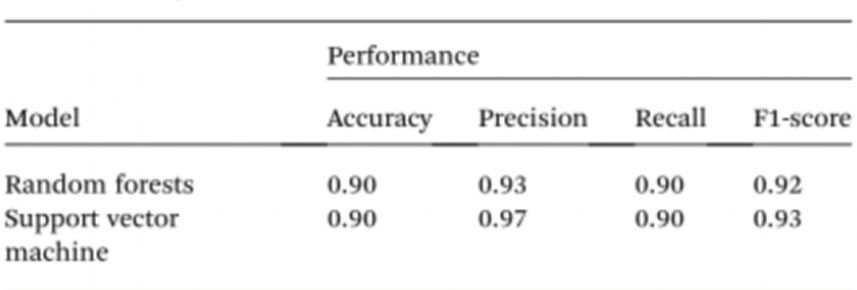
Aby jeszcze bardziej uprościć testowanie próbek bez użycia spektrofotometrów, naukowcy wykorzystali sztuczną inteligencję (AI) do interpretacji koloru roztworu i rozróżnienia osób normalnych i rakowych.Biorąc to pod uwagę, wykorzystano widzenie komputerowe do przełożenia koloru roztworu Cyst/AuNPs na normalne DNA (fioletowe) lub rakowe DNA (czerwone) przy użyciu obrazów 96-dołkowych płytek wykonanych aparatem w telefonie komórkowym.Sztuczna inteligencja może obniżyć koszty i poprawić dostępność w interpretacji koloru roztworów nanocząstek i to bez użycia jakichkolwiek akcesoriów do smartfonów ze sprzętem optycznym.Na koniec przeszkolono dwa modele uczenia maszynowego, w tym Random Forest (RF) i Support Vector Machine (SVM), aby skonstruować modele.zarówno modele RF, jak i SVM poprawnie sklasyfikowały próbki jako dodatnie i ujemne z dokładnością 90,0%.Sugeruje to, że wykorzystanie sztucznej inteligencji w bioczujnikach opartych na telefonach komórkowych jest całkiem możliwe.
Rycina 3.(a) Klasa docelowa roztworu zarejestrowana podczas przygotowania próbki do etapu akwizycji obrazu.(b) Przykładowe zdjęcie zrobione podczas etapu akwizycji obrazu.( c ) Intensywność koloru roztworu cysty / AuNPs w każdym dołku 96-dołkowej płytki wyekstrahowanej z obrazu ( b ).
Korzystając z Cyst/AuNP, naukowcy z powodzeniem opracowali prostą platformę detekcyjną do wykrywania krajobrazu metylacji oraz czujnik zdolny do odróżnienia normalnego DNA od DNA nowotworu przy użyciu prawdziwych próbek krwi do badań przesiewowych w kierunku białaczki.Opracowany czujnik wykazał, że DNA wyekstrahowane z prawdziwych próbek krwi było w stanie szybko i tanio wykryć niewielkie ilości DNA raka (3 nM) u pacjentów z białaczką w ciągu 15 minut i wykazał dokładność na poziomie 95,3%.Aby jeszcze bardziej uprościć testowanie próbek poprzez wyeliminowanie potrzeby stosowania spektrofotometru, wykorzystano uczenie maszynowe do interpretacji koloru roztworu i rozróżnienia osób zdrowych od osób z rakiem za pomocą zdjęcia wykonanego telefonem komórkowym, a także udało się osiągnąć dokładność na poziomie 90,0%.
Numer referencyjny: DOI: 10.1039/d2ra05725e
Czas postu: 18-02-2023
 中文网站
中文网站